안녕하세요. 오늘은 의료 영상 분할 핵심 모델이라고 할 수 있는 UNet의 변형 구조인 UNet++에 대해서 소개하도록 하겠습니다.
Background
의료 영상 분할 (medical image segmentation)는 현대 의료 기술에서 중요한 분야로 자리잡고 있습니다. 이는 질병의 조기 발견, 정확한 진단, 효과적인 치료 계획 수립 등에 필수적인 역할을 수행합니다. 특히 컴퓨터 비전과 딥러닝 기술의 발전으로, 의료 영상 분석에 인공지능을 활용한 연구가 활발히 진행되고 있습니다.
대표적인 의료 영상 분석 기술인 U-Net은 바이오메디컬 영역에서 뛰어난 성능을 보이며 많은 연구자들로부터 인정을 받아왔습니다. 그러나 최근 의료 영상이 더욱 복잡하고 정교해짐에 따라, 기존 U-Net 구조만으로는 미세한 병변과 구조물의 세부 사항을 충분히 정확하게 세분화하는 데 한계가 있었습니다.
이러한 배경에서, U-Net을 기반으로 하되 기존 구조의 한계를 뛰어넘어 정확성과 세부 표현력을 극대화한 'UNet++' 아키텍처가 등장했습니다. UNet++는 기존 U-Net의 기본 아이디어를 유지하면서도 인코더와 디코더 사이를 잇는 연결 구조(skip connection)를 중첩된 형태로 재설계하고, 깊은 지도학습(deep supervision)을 도입하여 성능을 향상시켰습니다. 본 논문의 기여는 다음과 같이 정리할 수 있습니다.
- UNet++는 인코더와 디코더 사이의 연결 구조를 중첩되고 밀집된 형태로 새롭게 설계함으로써 두 네트워크 간의 의미론적 간극(semantic gap)을 효과적으로 축소
- UNet++는 여러 단계에서 출력되는 중간 결과에 깊은 지도학습을 적용하여, 각 단계의 특징 맵을 세분화 작업에 최적화된 상태로 학습
- 폐결절, 결장 용종, 간, 세포핵 등 서로 다른 특성과 크기를 가진 의료 영상 데이터 세트에서 UNet++는 기존 U-Net 및 Wide U-Net 대비 평균 IoU 기준 각각 3.9점과 3.4점의 유의미한 성능 향상
Proposed Method: UNet++
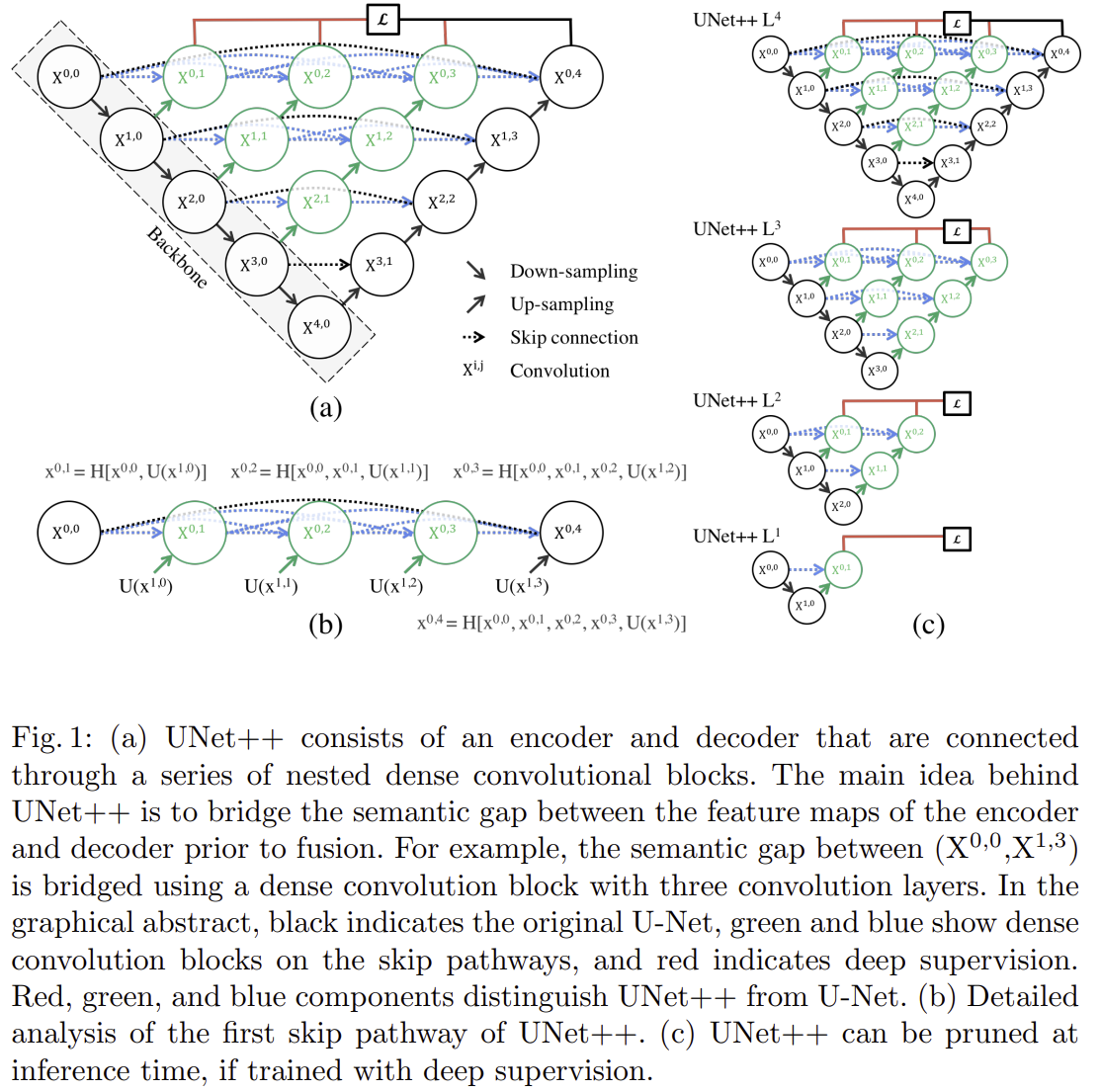
1) Re-designed skip pathways
그림 1은 UNet++의 전체적인 구조를 보여주고 있습니다. 의료 영상에서 질병이나 병변을 정확히 찾으려면, 미세한 세부사항을 놓치지 않고 명확히 잡아내는 것이 필수입니다. 기존에 널리 쓰이던 U-Net은 인코더(encoder)에서 얻은 특징 맵을 디코더(decoder)로 곧바로 전달하여 정보를 복원하는 구조를 사용했습니다. 하지만 이 방식은 인코더와 디코더 사이에서 특징 맵이 서로 다른 의미 수준(semantic level)을 가지기 때문에, 미세한 세부 정보를 포착하는 데 한계가 있었습니다.
이러한 문제를 해결하기 위해 UNet++는 스킵 연결(skip pathways)을 완전히 새롭게 재설계했습니다. UNet++의 재설계된 스킵 연결은 단순히 인코더에서 디코더로 정보를 바로 전달하지 않습니다. 대신 인코더에서 나온 특징 맵을 여러 개의 중첩된 밀집 컨볼루션 블록(nested dense convolution blocks)을 통과시켜, 특징을 더욱 정교하고 풍부하게 만들어줍니다. 이 과정에서 특징 맵들은 단계적으로 더 세부적인 정보를 얻고, 의미적(semantic)으로 디코더의 특징 맵과 유사한 상태가 되어 전달됩니다. 그 결과, UNet++는 의미적으로 인코더와 디코더 사이의 차이(semantic gap)를 줄이고, 더 세밀한 병변과 구조물의 표현을 가능하게 합니다.
스킵 연결을 통과하는 각 노드에서 생성된 특징 맵(feature map)을 $x^{i, j}$라고 할 때 이 과정을 수식으로 나타내면 다음과 같습니다.
$$x^{i, j} = \begin{cases} H(x^{i - 1, j}), &j = 0 \\ H(\left[ [x^{i, k}]_{k = 0}^{j - 1}, \mathcal{U}(x^{i + 1, j - 1}) \right]), &j > 0 \end{cases}$$
위 수식의 각 기호는 다음과 같은 의미를 갖습니다.
- $x^{i, j}$: 스킵 연결 내 노드 $X^{i, j}$ 에서 출력된 특징 맵으로 $i$ 는 인코더(encoder)의 다운샘플링(down-sampling) 단계, $j$ 는 각 단계 내 밀집 컨볼루션 블록(dense convolution block)의 컨볼루션 층(layer) 인덱스를 의미
- $H(\cdot)$: 컨볼루션(convolution) 연산과 활성화(activation) 함수로 이루어진 단일 연산 블록
- $\mathcal{U}(\cdot)$: 업샘플링(up-sampling) 연산
- $[\cdot]$: 특징 맵을 연결(concatenate)하는 연산
이러한 과정을 통해, UNet++의 스킵 연결은 각 노드에서 이전 단계와 하위 단계의 특징을 점진적으로 결합하여, 디코더에 전달될 때까지 점차적으로 의미론적으로 풍부한 특징 맵을 생성합니다. 또한, 이 수식 표현은 UNet++가 인코더의 저수준 특징(low-level features)을 디코더의 고수준 특징(high-level features)과 보다 의미적으로 일관성 있게 결합할 수 있도록 설계된 원리를 명확히 보여줍니다.
2) Deep Supervision
UNet++의 또 다른 중요한 특징은 깊은 지도학습(Deep Supervision)입니다. 기존의 의료 영상 세분화 모델은 대개 최종 출력 단계에서만 손실(loss)을 계산하고 이를 기반으로 네트워크를 훈련합니다. 하지만 이 방식은 중간 과정에서 생성된 특징 맵(feature maps)이 실제 세분화 작업에 얼마나 잘 기여하고 있는지에 대해 충분한 피드백을 제공하지 못합니다.
UNet++는 이 문제를 해결하기 위해 중간 단계에서도 지도학습을 수행하는 깊은 지도학습 방식을 사용합니다. 구체적으로, UNet++는 여러 개의 의미적 수준(semantic levels)을 갖는 중간 단계에서도 각각 독립적으로 세분화 결과를 생성하며, 이 모든 중간 결과에 대해 별도의 손실을 계산하여 훈련합니다. 이 과정에서 손실함수로는 binary cross-entropy loss와 dice coefficient loss의 합으로 정의되었습니다. 이를 수식으로 표현하면 다음과 같습니다.
$$\mathcal{L} (Y, \hat{Y}) = -\frac{1}{N} \sum_{b = 1}^{N} \left( \frac{1}{2} \cdot Y_{b} \cdot \log (\hat{Y}_{b}) + \frac{2 \cdot Y_{b} \cdot \hat{Y}_{b}}{Y_{b} + \hat{Y}_{b} } \right)$$
Experiment Results
1) Dataset Description
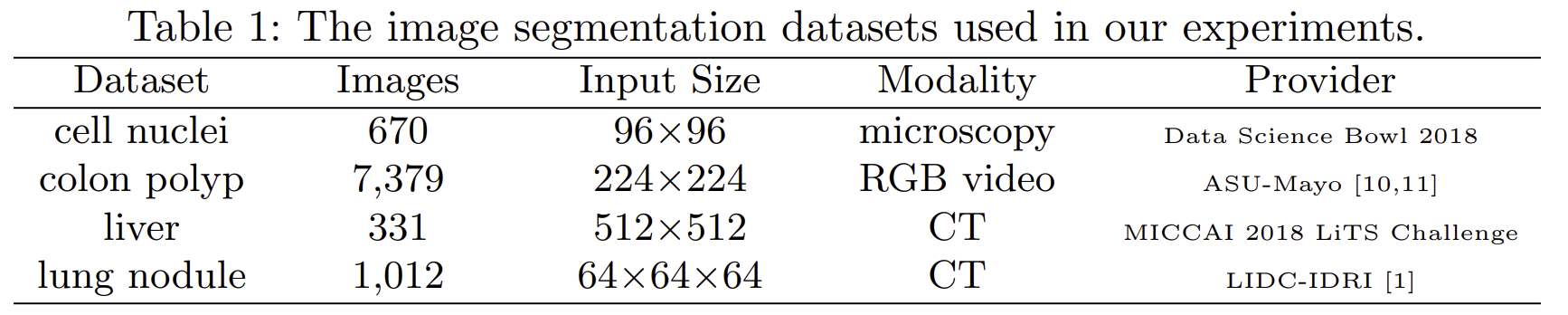
본 논문에서 사용한 데이터셋 종류는 표 1에서 볼 수 있습니다.
- 세포핵(Cell Nuclei) 데이터셋: 세포핵 데이터셋은 총 670장의 현미경 영상으로 구성되어 있으며, 각 영상의 크기는 96×96 픽셀입니다. 이 데이터는 2018년 Data Science Bowl에서 제공한 것으로, 세포핵의 형태와 크기가 다양하고 겹쳐 있는 경우도 많아 정확한 경계선을 찾아내는 세밀한 세분화(segmentation) 작업이 요구됩니다.
- 결장 용종(Colon Polyp) 데이터셋: 결장 용종 데이터셋은 총 7,379장의 RGB 영상으로 이루어진 내시경(colonoscopy) 데이터입니다. 각 영상 크기는 224×224 픽셀로, Arizona State University와 Mayo Clinic의 공동 연구를 통해 제공되었습니다. 용종은 크기와 형태가 매우 다양하며 영상마다 크게 달라지기 때문에, 일관된 성능을 유지하는 세분화 모델이 필수적입니다.
- 간(Liver) 데이터셋: 간 데이터셋은 복부 CT 영상 331장으로 구성되어 있으며, 영상의 크기는 512×512 픽셀입니다. MICCAI 2018 LiTS 챌린지에서 제공된 이 데이터셋은 간 조직과 간 내 병변을 명확하게 구별하는 정밀한 세분화를 요구합니다. 병변과 정상 조직 간의 경계가 불분명한 경우가 많아 높은 난이도의 세분화 작업으로 평가받습니다.
- 폐 결절(Lung Nodule) 데이터셋: 폐결절 데이터셋은 1,012장의 3차원(64×64×64 픽셀) 저선량 흉부 CT 영상으로 구성되었습니다. 이 데이터셋은 LIDC-IDRI에서 제공되었으며, 폐 내 미세 결절을 탐지하고 결절 주변의 미세한 스피큘레이션(spiculation) 패턴을 정확히 구분하는 작업을 목표로 합니다. 폐결절의 세분화 정확도는 결절의 악성 여부 판단에 매우 중요하므로 의료 영상 분석 분야에서 큰 주목을 받고 있습니다.
2) Implementation details
모든 실험에서는 Dice Coefficient와 IoU(Intersection over Union)를 사용하여 성능을 평가했습니다. 또한 모델의 과적합을 방지하기 위해 검증세트에서 조기 종료 기법을 적용하였습니다.
모델 학습은 Adam 최적화함수를 이용했고, 학습률은 $3 \times 10^{-4}$ 로 설정하여 최적의 학습 속도와 안정성을 유지했습니다. 네트워크의 인코더 및 디코더 구성은 Original U-Net을 기반으로 하였으며, 스킵 연결에서의 모든 컨볼루션 층은 커널 크기가 $3 \times 3$ (폐결절 데이터는 $3 \times 3 \times 3$)이고, 층마다 사용되는 컨볼루션 필터의 개수는 피라미드 수준에 따라 $32 \times 2^{i}$로 증가하도록 구성되었습니다.
특히 UNet++의 중요한 특징 중 하나인 깊은 지도학습(Deep Supervision)을 적용하기 위해, 네트워크의 여러 중간 단계에서 생성되는 특징 맵을 각각 독립적으로 세분화 출력으로 변환할 수 있도록 $1 \times 1$ 컨볼루션 층과 시그모이드(sigmoid) 활성화 함수를 추가했습니다. 이를 통해 UNet++는 하나의 입력 이미지에서 동시에 여러 개의 세분화 맵을 생성하며, 이 결과들을 평균화하여 최종 세분화 맵을 얻는 방식을 취합니다.
3) Baseline models

표 2는 본 논문에서 성능을 비교하는 baseline model들의 셋팅입니다. UNet++의 성능을 객관적으로 평가하기 위해 두 가지 기준 모델(baseline)을 사용하여 성능을 비교하였습니다. 선정된 두 모델은 Original U-Net과 Wide U-Net입니다.
- Original UNet: 의료 영상 세분화 분야에서 가장 널리 사용되고 있는 표준 모델로, 인코더-디코더 구조와 스킵 연결(skip connections)을 처음으로 의료 영상 분석에 효과적으로 적용한 모델입니다. 이 모델은 의료 영상 분석 연구의 표준적인 성능 평가 기준으로 사용되며, UNet++의 직접적인 비교 대상으로서 모델 개선의 기준점이 됩니다.
- Wide UNet: Original U-Net의 변형 모델로, 기본적인 구조는 동일하지만, 네트워크의 너비(width)를 증가시켜 컨볼루션 필터의 개수를 늘린 모델입니다. 이 모델을 선택한 이유는 단순히 UNet++의 성능 향상이 네트워크의 파라미터 수 증가 때문인지, 아니면 모델 구조 자체의 혁신으로 인한 것인지 확인하기 위해서였습니다. 즉, 파라미터 수를 UNet++와 비슷하게 맞추면서도 구조적 특징만 다르게 설정하여 공정한 비교가 가능하도록 하였습니다.
4) Results
4-1) Quantitative Results
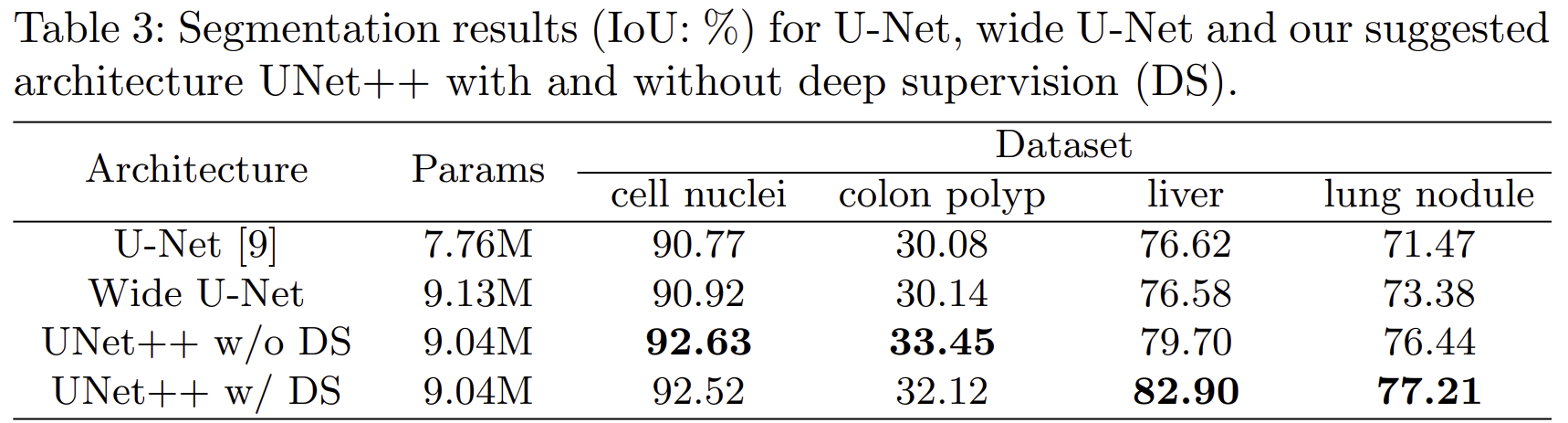
표 3은 전체적인 실험결과를 보여주고 있습니다. 본 논문에서는 기존 U-Net, Wide U-Net과 새롭게 제안된 UNet++ 모델(깊은 지도학습 유무)을 세포핵, 결장 용종, 간, 폐결절의 네 가지 의료 영상 데이터셋을 이용해 성능을 비교하였습니다.
- 세포핵 데이터셋에서는 UNet++ (Deep Supervision 없음)가 IoU 92.63%로 가장 높은 성능을 보였으며, Deep Supervision의 추가 효과는 미미했습니다.
- 결장 용종 데이터셋에서도 UNet++ (Deep Supervision 없음)가 33.45%로 가장 우수한 성능을 나타냈습니다.
- 간 데이터셋에서는 UNet++ (Deep Supervision 있음)가 82.90%의 압도적인 성능을 보이며 Deep Supervision의 효과가 뚜렷하게 나타났습니다.
- 폐결절 데이터셋 역시 UNet++ (Deep Supervision 있음)가 IoU 77.21%로 가장 우수했으며, Deep Supervision을 통한 성능 개선이 확인되었습니다.
전반적으로 UNet++는 기존 U-Net 및 Wide U-Net에 비해 모든 데이터셋에서 뚜렷한 성능 개선을 보였으며, 특히 간과 폐결절 데이터셋에서 깊은 지도학습의 효과가 명확히 나타났습니다.
4-2) Qualitative Results
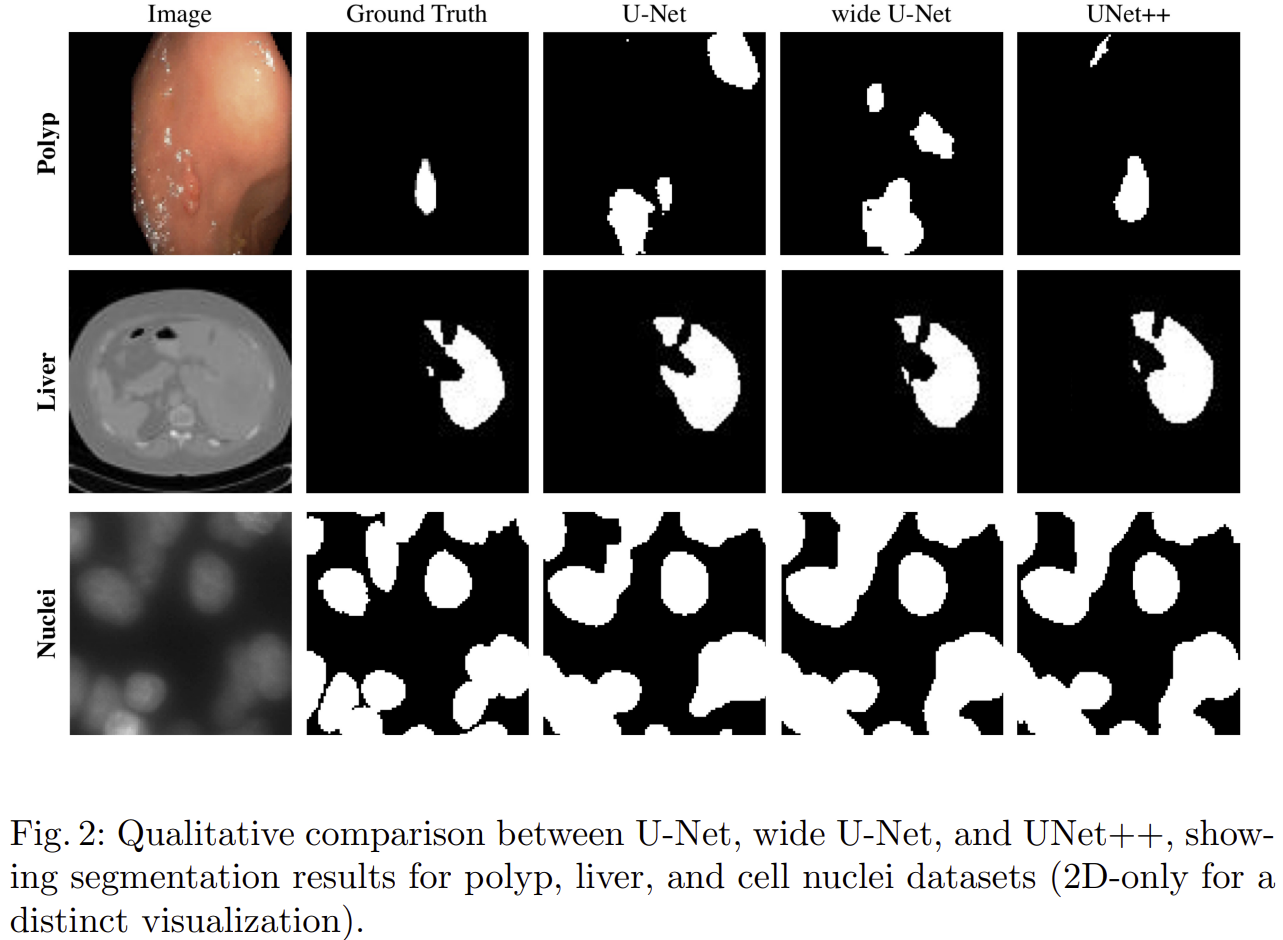
그림 2는 Qualtiative result를 보여주고 있습니다.
- 결장 용종 데이터셋에서 UNet++는 실제 정답(Ground Truth)과 거의 유사한 세분화를 수행하여 병변을 명확히 잡아낸 반면, 기존 U-Net 및 Wide U-Net은 주변의 불필요한 영역까지 오탐지(false detection)하는 모습을 보였습니다.
- 간 데이터셋에서 UNet++는 간 영역의 미세한 경계를 더 정교하게 표현하여 실제 정답에 가장 가까운 결과를 얻었습니다. 기존 U-Net과 Wide U-Net은 간 외부의 영역을 다소 부정확하게 포함하거나 일부 중요한 경계를 놓쳤습니다.
- 세포핵 데이터셋에서 UNet++는 세포핵의 경계를 뚜렷하게 구분하면서 각 세포를 보다 정확하게 분리한 반면, Original U-Net 및 Wide U-Net은 일부 세포가 서로 붙어서 제대로 분리되지 않는 현상을 보였습니다.
종합적으로, UNet++가 세 가지 데이터셋 모두에서 실제 정답에 가장 가까운 정교한 세분화 성능을 시각적으로 입증하였습니다.
5) Efficiency Analysis
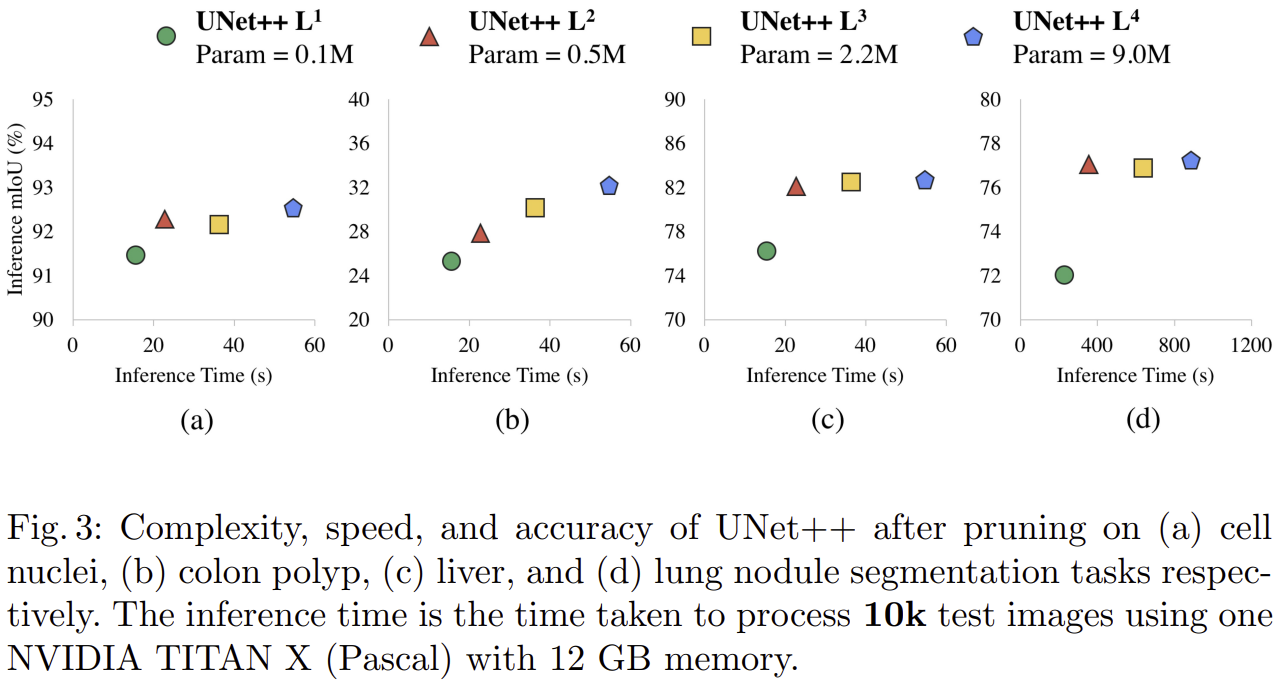
UNet++는 깊은 지도학습(Deep Supervision)을 이용해 필요에 따라 네트워크 구조를 축소(가지치기, pruning)할 수 있는 장점을 제공합니다. 위의 실험 결과는 서로 다른 수준으로 가지치기한 UNet++의 성능과 추론 속도(Inference Time)를 네 가지 의료 영상 세분화 데이터셋에 대해 분석한 것입니다.
- 세포핵 데이터셋에서는 가장 간소화된 UNet++ L¹ (0.1M)도 90% 이상의 IoU를 유지하며 빠른 추론 속도를 보였으며, 모델의 크기를 증가시킬수록 정확도가 다소 향상되었습니다.
- 결장 용종 데이터셋의 경우 모델을 간소화할수록 성능 저하가 다소 크게 나타났으나, L³ (2.2M) 이상의 모델에서는 성능이 일정 수준 안정화되었습니다.
- 간 데이터셋 에서는 간소화된 모델(L¹, L²)이 상대적으로 낮은 정확도를 보였지만, 모델 크기가 증가할수록 정확도가 뚜렷이 개선되었습니다. L³ 수준 이상에서 좋은 정확도와 속도의 균형점을 보였습니다.
- 폐결절 데이터셋에서는 가장 작은 모델(L¹)의 성능 저하가 가장 컸으나, 중간 수준 이상의 모델(L², L³)에서는 추론 시간이 상대적으로 적게 증가하면서 정확도가 안정적으로 유지되었습니다.
종합적으로, 모델 가지치기를 통해 UNet++는 의료 영상 세분화 작업에서 높은 정확도를 유지하면서도 현장에 필요한 속도와 연산 효율성을 최적화할 수 있음을 보여줬습니다.
Conclusion
본 논문에서 제안한 UNet++는 기존 의료 영상 세분화 모델인 Original U-Net을 기반으로 하면서도, 인코더와 디코더 사이의 스킵 연결을 중첩된 밀집 연결(Nested Dense Skip Pathways)로 재설계하고, 깊은 지도학습(Deep Supervision)을 도입하여 성능을 혁신적으로 향상시킨 모델입니다.
실험을 통해 UNet++는 세포핵, 결장 용종, 간, 폐결절 등 다양한 특성의 의료 영상 데이터셋에서 일관되게 기존 모델들(U-Net, Wide U-Net)보다 뛰어난 성능을 보였습니다. 특히 병변의 형태와 크기가 다양하고 경계가 복잡한 간과 폐결절 데이터셋에서는 깊은 지도학습의 효과가 명확히 드러났습니다. 반면 상대적으로 단순한 세포핵 및 결장 용종 데이터에서는 오히려 깊은 지도학습의 장점이 제한적인 것으로 나타나, 데이터 특성에 따른 모델 구조의 적합성도 중요한 고려 요소임을 확인하였습니다.
또한, UNet++는 모델 가지치기(pruning)를 통해 정확성과 연산 효율성 사이의 균형을 효과적으로 맞출 수 있어, 실제 의료 현장에서의 활용성 또한 뛰어납니다.
결론적으로 UNet++는 의미론적 간극 축소, 다층적 학습 구조, 유연한 효율성 조정 등 혁신적이고 실질적인 기능들을 통해 의료 영상 세분화 분야에서의 새로운 기준을 제시했다고 평가할 수 있습니다.
'Paper Review' 카테고리의 다른 글
| Deformable Convolutional Networks (ICCV2017) (0) | 2025.04.05 |
|---|---|
| MISSFormer: An Effective Medical Image Segmentation Transformer (IEEE TMI2023) (0) | 2025.04.04 |
| Multiscale Vision Transformers (ICCV2021) (0) | 2025.03.28 |
| Segmenter: Transformer for Semantic Segmentation (ICCV2021) (0) | 2025.03.25 |
| Inception Transformer (NIPS2022) (0) | 2025.03.09 |
안녕하세요. 오늘은 의료 영상 분할 핵심 모델이라고 할 수 있는 UNet의 변형 구조인 UNet++에 대해서 소개하도록 하겠습니다.
Background
의료 영상 분할 (medical image segmentation)는 현대 의료 기술에서 중요한 분야로 자리잡고 있습니다. 이는 질병의 조기 발견, 정확한 진단, 효과적인 치료 계획 수립 등에 필수적인 역할을 수행합니다. 특히 컴퓨터 비전과 딥러닝 기술의 발전으로, 의료 영상 분석에 인공지능을 활용한 연구가 활발히 진행되고 있습니다.
대표적인 의료 영상 분석 기술인 U-Net은 바이오메디컬 영역에서 뛰어난 성능을 보이며 많은 연구자들로부터 인정을 받아왔습니다. 그러나 최근 의료 영상이 더욱 복잡하고 정교해짐에 따라, 기존 U-Net 구조만으로는 미세한 병변과 구조물의 세부 사항을 충분히 정확하게 세분화하는 데 한계가 있었습니다.
이러한 배경에서, U-Net을 기반으로 하되 기존 구조의 한계를 뛰어넘어 정확성과 세부 표현력을 극대화한 'UNet++' 아키텍처가 등장했습니다. UNet++는 기존 U-Net의 기본 아이디어를 유지하면서도 인코더와 디코더 사이를 잇는 연결 구조(skip connection)를 중첩된 형태로 재설계하고, 깊은 지도학습(deep supervision)을 도입하여 성능을 향상시켰습니다. 본 논문의 기여는 다음과 같이 정리할 수 있습니다.
- UNet++는 인코더와 디코더 사이의 연결 구조를 중첩되고 밀집된 형태로 새롭게 설계함으로써 두 네트워크 간의 의미론적 간극(semantic gap)을 효과적으로 축소
- UNet++는 여러 단계에서 출력되는 중간 결과에 깊은 지도학습을 적용하여, 각 단계의 특징 맵을 세분화 작업에 최적화된 상태로 학습
- 폐결절, 결장 용종, 간, 세포핵 등 서로 다른 특성과 크기를 가진 의료 영상 데이터 세트에서 UNet++는 기존 U-Net 및 Wide U-Net 대비 평균 IoU 기준 각각 3.9점과 3.4점의 유의미한 성능 향상
Proposed Method: UNet++

1) Re-designed skip pathways
그림 1은 UNet++의 전체적인 구조를 보여주고 있습니다. 의료 영상에서 질병이나 병변을 정확히 찾으려면, 미세한 세부사항을 놓치지 않고 명확히 잡아내는 것이 필수입니다. 기존에 널리 쓰이던 U-Net은 인코더(encoder)에서 얻은 특징 맵을 디코더(decoder)로 곧바로 전달하여 정보를 복원하는 구조를 사용했습니다. 하지만 이 방식은 인코더와 디코더 사이에서 특징 맵이 서로 다른 의미 수준(semantic level)을 가지기 때문에, 미세한 세부 정보를 포착하는 데 한계가 있었습니다.
이러한 문제를 해결하기 위해 UNet++는 스킵 연결(skip pathways)을 완전히 새롭게 재설계했습니다. UNet++의 재설계된 스킵 연결은 단순히 인코더에서 디코더로 정보를 바로 전달하지 않습니다. 대신 인코더에서 나온 특징 맵을 여러 개의 중첩된 밀집 컨볼루션 블록(nested dense convolution blocks)을 통과시켜, 특징을 더욱 정교하고 풍부하게 만들어줍니다. 이 과정에서 특징 맵들은 단계적으로 더 세부적인 정보를 얻고, 의미적(semantic)으로 디코더의 특징 맵과 유사한 상태가 되어 전달됩니다. 그 결과, UNet++는 의미적으로 인코더와 디코더 사이의 차이(semantic gap)를 줄이고, 더 세밀한 병변과 구조물의 표현을 가능하게 합니다.
스킵 연결을 통과하는 각 노드에서 생성된 특징 맵(feature map)을 $x^{i, j}$라고 할 때 이 과정을 수식으로 나타내면 다음과 같습니다.
$$x^{i, j} = \begin{cases} H(x^{i - 1, j}), &j = 0 \\ H(\left[ [x^{i, k}]_{k = 0}^{j - 1}, \mathcal{U}(x^{i + 1, j - 1}) \right]), &j > 0 \end{cases}$$
위 수식의 각 기호는 다음과 같은 의미를 갖습니다.
- $x^{i, j}$: 스킵 연결 내 노드 $X^{i, j}$ 에서 출력된 특징 맵으로 $i$ 는 인코더(encoder)의 다운샘플링(down-sampling) 단계, $j$ 는 각 단계 내 밀집 컨볼루션 블록(dense convolution block)의 컨볼루션 층(layer) 인덱스를 의미
- $H(\cdot)$: 컨볼루션(convolution) 연산과 활성화(activation) 함수로 이루어진 단일 연산 블록
- $\mathcal{U}(\cdot)$: 업샘플링(up-sampling) 연산
- $[\cdot]$: 특징 맵을 연결(concatenate)하는 연산
이러한 과정을 통해, UNet++의 스킵 연결은 각 노드에서 이전 단계와 하위 단계의 특징을 점진적으로 결합하여, 디코더에 전달될 때까지 점차적으로 의미론적으로 풍부한 특징 맵을 생성합니다. 또한, 이 수식 표현은 UNet++가 인코더의 저수준 특징(low-level features)을 디코더의 고수준 특징(high-level features)과 보다 의미적으로 일관성 있게 결합할 수 있도록 설계된 원리를 명확히 보여줍니다.
2) Deep Supervision
UNet++의 또 다른 중요한 특징은 깊은 지도학습(Deep Supervision)입니다. 기존의 의료 영상 세분화 모델은 대개 최종 출력 단계에서만 손실(loss)을 계산하고 이를 기반으로 네트워크를 훈련합니다. 하지만 이 방식은 중간 과정에서 생성된 특징 맵(feature maps)이 실제 세분화 작업에 얼마나 잘 기여하고 있는지에 대해 충분한 피드백을 제공하지 못합니다.
UNet++는 이 문제를 해결하기 위해 중간 단계에서도 지도학습을 수행하는 깊은 지도학습 방식을 사용합니다. 구체적으로, UNet++는 여러 개의 의미적 수준(semantic levels)을 갖는 중간 단계에서도 각각 독립적으로 세분화 결과를 생성하며, 이 모든 중간 결과에 대해 별도의 손실을 계산하여 훈련합니다. 이 과정에서 손실함수로는 binary cross-entropy loss와 dice coefficient loss의 합으로 정의되었습니다. 이를 수식으로 표현하면 다음과 같습니다.
$$\mathcal{L} (Y, \hat{Y}) = -\frac{1}{N} \sum_{b = 1}^{N} \left( \frac{1}{2} \cdot Y_{b} \cdot \log (\hat{Y}_{b}) + \frac{2 \cdot Y_{b} \cdot \hat{Y}_{b}}{Y_{b} + \hat{Y}_{b} } \right)$$
Experiment Results
1) Dataset Description

본 논문에서 사용한 데이터셋 종류는 표 1에서 볼 수 있습니다.
- 세포핵(Cell Nuclei) 데이터셋: 세포핵 데이터셋은 총 670장의 현미경 영상으로 구성되어 있으며, 각 영상의 크기는 96×96 픽셀입니다. 이 데이터는 2018년 Data Science Bowl에서 제공한 것으로, 세포핵의 형태와 크기가 다양하고 겹쳐 있는 경우도 많아 정확한 경계선을 찾아내는 세밀한 세분화(segmentation) 작업이 요구됩니다.
- 결장 용종(Colon Polyp) 데이터셋: 결장 용종 데이터셋은 총 7,379장의 RGB 영상으로 이루어진 내시경(colonoscopy) 데이터입니다. 각 영상 크기는 224×224 픽셀로, Arizona State University와 Mayo Clinic의 공동 연구를 통해 제공되었습니다. 용종은 크기와 형태가 매우 다양하며 영상마다 크게 달라지기 때문에, 일관된 성능을 유지하는 세분화 모델이 필수적입니다.
- 간(Liver) 데이터셋: 간 데이터셋은 복부 CT 영상 331장으로 구성되어 있으며, 영상의 크기는 512×512 픽셀입니다. MICCAI 2018 LiTS 챌린지에서 제공된 이 데이터셋은 간 조직과 간 내 병변을 명확하게 구별하는 정밀한 세분화를 요구합니다. 병변과 정상 조직 간의 경계가 불분명한 경우가 많아 높은 난이도의 세분화 작업으로 평가받습니다.
- 폐 결절(Lung Nodule) 데이터셋: 폐결절 데이터셋은 1,012장의 3차원(64×64×64 픽셀) 저선량 흉부 CT 영상으로 구성되었습니다. 이 데이터셋은 LIDC-IDRI에서 제공되었으며, 폐 내 미세 결절을 탐지하고 결절 주변의 미세한 스피큘레이션(spiculation) 패턴을 정확히 구분하는 작업을 목표로 합니다. 폐결절의 세분화 정확도는 결절의 악성 여부 판단에 매우 중요하므로 의료 영상 분석 분야에서 큰 주목을 받고 있습니다.
2) Implementation details
모든 실험에서는 Dice Coefficient와 IoU(Intersection over Union)를 사용하여 성능을 평가했습니다. 또한 모델의 과적합을 방지하기 위해 검증세트에서 조기 종료 기법을 적용하였습니다.
모델 학습은 Adam 최적화함수를 이용했고, 학습률은 $3 \times 10^{-4}$ 로 설정하여 최적의 학습 속도와 안정성을 유지했습니다. 네트워크의 인코더 및 디코더 구성은 Original U-Net을 기반으로 하였으며, 스킵 연결에서의 모든 컨볼루션 층은 커널 크기가 $3 \times 3$ (폐결절 데이터는 $3 \times 3 \times 3$)이고, 층마다 사용되는 컨볼루션 필터의 개수는 피라미드 수준에 따라 $32 \times 2^{i}$로 증가하도록 구성되었습니다.
특히 UNet++의 중요한 특징 중 하나인 깊은 지도학습(Deep Supervision)을 적용하기 위해, 네트워크의 여러 중간 단계에서 생성되는 특징 맵을 각각 독립적으로 세분화 출력으로 변환할 수 있도록 $1 \times 1$ 컨볼루션 층과 시그모이드(sigmoid) 활성화 함수를 추가했습니다. 이를 통해 UNet++는 하나의 입력 이미지에서 동시에 여러 개의 세분화 맵을 생성하며, 이 결과들을 평균화하여 최종 세분화 맵을 얻는 방식을 취합니다.
3) Baseline models

표 2는 본 논문에서 성능을 비교하는 baseline model들의 셋팅입니다. UNet++의 성능을 객관적으로 평가하기 위해 두 가지 기준 모델(baseline)을 사용하여 성능을 비교하였습니다. 선정된 두 모델은 Original U-Net과 Wide U-Net입니다.
- Original UNet: 의료 영상 세분화 분야에서 가장 널리 사용되고 있는 표준 모델로, 인코더-디코더 구조와 스킵 연결(skip connections)을 처음으로 의료 영상 분석에 효과적으로 적용한 모델입니다. 이 모델은 의료 영상 분석 연구의 표준적인 성능 평가 기준으로 사용되며, UNet++의 직접적인 비교 대상으로서 모델 개선의 기준점이 됩니다.
- Wide UNet: Original U-Net의 변형 모델로, 기본적인 구조는 동일하지만, 네트워크의 너비(width)를 증가시켜 컨볼루션 필터의 개수를 늘린 모델입니다. 이 모델을 선택한 이유는 단순히 UNet++의 성능 향상이 네트워크의 파라미터 수 증가 때문인지, 아니면 모델 구조 자체의 혁신으로 인한 것인지 확인하기 위해서였습니다. 즉, 파라미터 수를 UNet++와 비슷하게 맞추면서도 구조적 특징만 다르게 설정하여 공정한 비교가 가능하도록 하였습니다.
4) Results
4-1) Quantitative Results

표 3은 전체적인 실험결과를 보여주고 있습니다. 본 논문에서는 기존 U-Net, Wide U-Net과 새롭게 제안된 UNet++ 모델(깊은 지도학습 유무)을 세포핵, 결장 용종, 간, 폐결절의 네 가지 의료 영상 데이터셋을 이용해 성능을 비교하였습니다.
- 세포핵 데이터셋에서는 UNet++ (Deep Supervision 없음)가 IoU 92.63%로 가장 높은 성능을 보였으며, Deep Supervision의 추가 효과는 미미했습니다.
- 결장 용종 데이터셋에서도 UNet++ (Deep Supervision 없음)가 33.45%로 가장 우수한 성능을 나타냈습니다.
- 간 데이터셋에서는 UNet++ (Deep Supervision 있음)가 82.90%의 압도적인 성능을 보이며 Deep Supervision의 효과가 뚜렷하게 나타났습니다.
- 폐결절 데이터셋 역시 UNet++ (Deep Supervision 있음)가 IoU 77.21%로 가장 우수했으며, Deep Supervision을 통한 성능 개선이 확인되었습니다.
전반적으로 UNet++는 기존 U-Net 및 Wide U-Net에 비해 모든 데이터셋에서 뚜렷한 성능 개선을 보였으며, 특히 간과 폐결절 데이터셋에서 깊은 지도학습의 효과가 명확히 나타났습니다.
4-2) Qualitative Results

그림 2는 Qualtiative result를 보여주고 있습니다.
- 결장 용종 데이터셋에서 UNet++는 실제 정답(Ground Truth)과 거의 유사한 세분화를 수행하여 병변을 명확히 잡아낸 반면, 기존 U-Net 및 Wide U-Net은 주변의 불필요한 영역까지 오탐지(false detection)하는 모습을 보였습니다.
- 간 데이터셋에서 UNet++는 간 영역의 미세한 경계를 더 정교하게 표현하여 실제 정답에 가장 가까운 결과를 얻었습니다. 기존 U-Net과 Wide U-Net은 간 외부의 영역을 다소 부정확하게 포함하거나 일부 중요한 경계를 놓쳤습니다.
- 세포핵 데이터셋에서 UNet++는 세포핵의 경계를 뚜렷하게 구분하면서 각 세포를 보다 정확하게 분리한 반면, Original U-Net 및 Wide U-Net은 일부 세포가 서로 붙어서 제대로 분리되지 않는 현상을 보였습니다.
종합적으로, UNet++가 세 가지 데이터셋 모두에서 실제 정답에 가장 가까운 정교한 세분화 성능을 시각적으로 입증하였습니다.
5) Efficiency Analysis

UNet++는 깊은 지도학습(Deep Supervision)을 이용해 필요에 따라 네트워크 구조를 축소(가지치기, pruning)할 수 있는 장점을 제공합니다. 위의 실험 결과는 서로 다른 수준으로 가지치기한 UNet++의 성능과 추론 속도(Inference Time)를 네 가지 의료 영상 세분화 데이터셋에 대해 분석한 것입니다.
- 세포핵 데이터셋에서는 가장 간소화된 UNet++ L¹ (0.1M)도 90% 이상의 IoU를 유지하며 빠른 추론 속도를 보였으며, 모델의 크기를 증가시킬수록 정확도가 다소 향상되었습니다.
- 결장 용종 데이터셋의 경우 모델을 간소화할수록 성능 저하가 다소 크게 나타났으나, L³ (2.2M) 이상의 모델에서는 성능이 일정 수준 안정화되었습니다.
- 간 데이터셋 에서는 간소화된 모델(L¹, L²)이 상대적으로 낮은 정확도를 보였지만, 모델 크기가 증가할수록 정확도가 뚜렷이 개선되었습니다. L³ 수준 이상에서 좋은 정확도와 속도의 균형점을 보였습니다.
- 폐결절 데이터셋에서는 가장 작은 모델(L¹)의 성능 저하가 가장 컸으나, 중간 수준 이상의 모델(L², L³)에서는 추론 시간이 상대적으로 적게 증가하면서 정확도가 안정적으로 유지되었습니다.
종합적으로, 모델 가지치기를 통해 UNet++는 의료 영상 세분화 작업에서 높은 정확도를 유지하면서도 현장에 필요한 속도와 연산 효율성을 최적화할 수 있음을 보여줬습니다.
Conclusion
본 논문에서 제안한 UNet++는 기존 의료 영상 세분화 모델인 Original U-Net을 기반으로 하면서도, 인코더와 디코더 사이의 스킵 연결을 중첩된 밀집 연결(Nested Dense Skip Pathways)로 재설계하고, 깊은 지도학습(Deep Supervision)을 도입하여 성능을 혁신적으로 향상시킨 모델입니다.
실험을 통해 UNet++는 세포핵, 결장 용종, 간, 폐결절 등 다양한 특성의 의료 영상 데이터셋에서 일관되게 기존 모델들(U-Net, Wide U-Net)보다 뛰어난 성능을 보였습니다. 특히 병변의 형태와 크기가 다양하고 경계가 복잡한 간과 폐결절 데이터셋에서는 깊은 지도학습의 효과가 명확히 드러났습니다. 반면 상대적으로 단순한 세포핵 및 결장 용종 데이터에서는 오히려 깊은 지도학습의 장점이 제한적인 것으로 나타나, 데이터 특성에 따른 모델 구조의 적합성도 중요한 고려 요소임을 확인하였습니다.
또한, UNet++는 모델 가지치기(pruning)를 통해 정확성과 연산 효율성 사이의 균형을 효과적으로 맞출 수 있어, 실제 의료 현장에서의 활용성 또한 뛰어납니다.
결론적으로 UNet++는 의미론적 간극 축소, 다층적 학습 구조, 유연한 효율성 조정 등 혁신적이고 실질적인 기능들을 통해 의료 영상 세분화 분야에서의 새로운 기준을 제시했다고 평가할 수 있습니다.
'Paper Review' 카테고리의 다른 글
| Deformable Convolutional Networks (ICCV2017) (0) | 2025.04.05 |
|---|---|
| MISSFormer: An Effective Medical Image Segmentation Transformer (IEEE TMI2023) (0) | 2025.04.04 |
| Multiscale Vision Transformers (ICCV2021) (0) | 2025.03.28 |
| Segmenter: Transformer for Semantic Segmentation (ICCV2021) (0) | 2025.03.25 |
| Inception Transformer (NIPS2022) (0) | 2025.03.09 |
